El establecimiento de modelos in vitro que imiten la invasión tumoral como parte del proceso metastásico es una parte fundamental de la investigación oncológica. La necesidad de incorporar múltiples tipos de células, análisis cinéticos a largo plazo, métodos para permitir la invasión 3D en la matriz circundante y la detección y el análisis apropiados ha dificultado la creación de nuevos modelos.
Autores
Brad Larson. Agilent Technologies, Inc. Winooski, VT, USA
Jan Seldin Greiner Bio-One North America, Inc. Monroe, NC, USA
Resumen
El establecimiento de modelos in vitro que imiten la invasión tumoral como parte del proceso metastásico es una parte fundamental de la investigación oncológica. La necesidad de incorporar múltiples tipos de células, análisis cinéticos a largo plazo, métodos para permitir la invasión 3D en la matriz circundante y la detección y el análisis apropiados ha dificultado la creación de nuevos modelos. El procedimiento descripto en esta nota de aplicación satisface estas necesidades mediante la inclusión de capacidades de imagen avanzadas que permiten la captura de múltiples imágenes en un rango de alturas z, utilizando canales de fluorescencia y campo claro de forma cinética. Las imágenes procesadas finales, después de la unión y la proyección z, permiten un análisis celular preciso para discernir las capacidades invasivas de las estructuras celulares 3D a lo largo del tiempo.
Introducción
El descubrimiento de fármacos oncológicos se ha enfrentado a múltiples desafíos a lo largo de los años. Dado que los cánceres desarrollan múltiples mutaciones durante la carcinogénesis, los enfoques dirigidos a mutaciones genéticas individuales comunes a muchas campañas de descubrimiento de fármacos tienen una eficacia limitada en su mayoría. Por el contrario, el avance de métodos novedosos que se centran en la inhibición del fenotipo invasivo de la metástasis ofrece un mayor potencial para una intervención significativa, particularmente debido al hecho de que la mayoría de los pacientes con cáncer mueren solo después de que se ha producido la metástasis. Para que este enfoque funcione, los modelos celulares in vitro utilizados en el descubrimiento temprano de fármacos deben imitar lo más cerca posible el complejo proceso metastásico. Los tumores in vivo existen como una masa tridimensional (3D) de múltiples tipos de células, incluidas las células cancerosas y del estroma. Por lo tanto, la incorporación de una estructura celular de tipo esferoide 3D que incluye tipos de células cocultivadas que forman un tumoroide proporciona un modelo más predictivo que el uso de células cancerosas individuales sembradas en microplacas. Otra limitación es la necesidad de experimentos cinéticos a largo plazo para capturar el proceso de invasión que normalmente requiere varios días. Por lo tanto, se necesita un control ambiental del ensayo de modo que la viabilidad celular del esferoide se mantenga durante este largo período y se evalúen correctamente los efectos putativos del fármaco. Finalmente, la única lectura adecuada para monitorear la invasión tumoral es la microscopía debido al pequeño tamaño del esferoide. Para ser efectivo en el descubrimiento de fármacos, el microscopio debe tener captura, procesamiento y análisis de imágenes automatizados para poder hacer frente a las múltiples condiciones de ensayo, compuestos probados e imágenes cinéticas tomadas. Este trabajo demostrará un procedimiento para la generación de estructuras tumoroides esferoidales en 3D, la creación de una matriz de invasión adecuada, el monitoreo automatizado basado en imágenes cinéticas y el análisis celular de imágenes apiladas en z capturadas de invasión tumoral.
En este estudio se utilizaron líneas celulares de glioblastoma multiforme (GBM) U-87 y LN-229, ya que han demostrado diferencias fenotípicas y capacidad metastásica.1 En particular, el gen PTEN supresor del crecimiento está mutado en las células U-87, pero funciona normalmente en las células LN-229. Además, la proteína fosfotransferasa del citomegalovirus humano UL 97 inhibe la elongación y la replicación del ADN y está ausente en las células U-87, pero está presente en las células LN-229.2 Esto respalda un patrón de crecimiento e invasión más agresivo para las células U-87. Ambos tipos de células se cocultivaron con fibroblastos para crear tumoroides 3D que representaban mejor las condiciones tumorales in vivo y se les permitió invadir a través de una matriz proteica.
La 17-alilamino-17-desmetoxigeldanamicina (17-AAG), conocida por inhibir la función de la proteína de choque térmico 90 (Hsp90), una proteína chaperona que estabiliza las proteínas necesarias para el crecimiento tumoral, se utilizó aquí para inhibir la posible invasión tumoral.3 Se utilizó la cuantificación de las imágenes cinéticas capturadas para caracterizar el potencial invasor de cultivos tumoroides inhibidos y no inhibidos.
Materiales y métodos
Materiales
Células
Las células GBM U-87 que expresan GFP fueron generosamente donadas por el Dr. Sachin Katyal (Universidad de Manitoba, Winnipeg, Manitoba, Canadá). Las células GBM LN-229 (número de pieza CRL-2611) se obtuvieron de ATCC (Manassas, VA). Se adquirieron fibroblastos dérmicos neonatales humanos que expresan RFP (número de pieza cAP-0008RFP) de Angio-Proteomie (Boston, MA).
Componentes experimentales
La matriz de membrana basal Matrigel, sin rojo de fenol (número de pieza 356237) se adquirió de Corning Life Sciences (Corning, NY). El inhibidor selectivo de Hsp90 17-AAG (número de pieza 1515) se obtuvo de Tocris Bioscience (Avonmouth, Bristol, Reino Unido). El tinte CellTracker Deep Red (número de pieza C34565) se adquirió de Thermo Fisher Scientific (Waltham, MA). Greiner Bio-One North America, Inc. (Monroe, NC) donó generosamente microplacas de 96 pocillos, repelentes de células, de poliestireno, de fondo redondo, transparentes y estériles con tapa (número de pieza 650979).
Lector multimodo de imágenes de células Agilent BioTek Cytation 5
Cytation 5 es un lector de microplacas multimodo modular combinado con un microscopio digital automatizado. La lectura de microplacas basada en filtro y monocromador está disponible, y el módulo de microscopía proporciona un aumento de hasta 60x en fluorescencia, campo claro, campo claro de color y contraste de fase. El instrumento puede realizar imágenes de fluorescencia en hasta cuatro canales en un solo paso. Con especial énfasis en los ensayos de células vivas, Cytation 5 presenta agitación, control de temperatura a 65°C, control de gas CO2/O2 e inyectores duales para ensayos cinéticos y está controlado por el software integrado de lector de microplacas y generador de imágenes Agilent BioTek Gen5, que también automatiza la captura de imágenes, análisis y procesamiento. El equipo se usó para monitorear cinéticamente la actividad tumoroide 3D durante el período de incubación.
Incubadora automatizada Agilent BioTek BioSpa 8
La incubadora automatizada BioSpa 8 conecta lectores o reproductores de imágenes Agilent BioTek con lavadoras y dispensadores Agilent BioTek para una automatización completa del flujo de trabajo de hasta ocho microplacas. Los niveles de temperatura, CO2/O2 y humedad se controlan y supervisan a través del software Agilent BioTek BioSpa para mantener un entorno ideal para los cultivos celulares durante todas las etapas experimentales. Las placas de prueba se incubaron en el BioSpa para mantener las condiciones atmosféricas adecuadas durante un período de siete días y se transfirieron automáticamente al Cytation 5 cada doce horas para obtener imágenes de campo claro y de fluorescencia.
Dispensador multimodo Agilent BioTek MultiFlo FX
El MultiFlo FX es un dispensador de reactivo modular y actualizable que puede tener hasta dos bombas perimétricas (dispensadores de 8 tubos), dos dispensadores de bomba de jeringa y una lavadora de tiras. Los colectores de jeringa y lavadora se pueden configurar para densidades de placa de 6 a 384 pocillos.
Métodos
Preparación celular y formación de tumoroides
Se recogieron células U-87 y fibroblastos preparados y se combinaron en una concentración final de 2,5 × 104 células/mL para cada tipo de célula en medio completo. Después de dispensar 100 μL de suspensión celular en los pocillos de la microplaca apropiados, la microplaca se incubó a 37°C/CO2 al 5 % durante 48 horas para permitir que las células se agregaran en tumoroides. Este proceso se repitió usando células LN-229 teñidas con el tinte CellTracker Deep Red y células de fibroblastos en las mismas concentraciones y volúmenes.
Preparación de la matriz de invasión
Una vez completada la formación del tumoroide, se extrajeron robóticamente 70 μl de medio completo de cada pocillo y la placa del tumoroide se colocó en hielo en un refrigerador durante cinco minutos para enfriar las células. A continuación, la matriz de Matrigel se descongeló en hielo. El 17-AAG se diluyó a una concentración 2x de 20 000 nM en medios de invasión y se usó para crear una titulación de ocho puntos de 20.000 nM a 0 nM usando diluciones en serie 1:4. Las titulaciones del inhibidor se diluyeron aún más a una concentración de 1x combinándolas con medios de invasión o matriz de Matrigel. Con la placa todavía en hielo, se agregaron 70 μL de matriz de Matrigel más el compuesto titulado a cada pocillo que contenía tumoroides, luego se cubrieron con 100 μL de medio de invasión que contenía el compuesto titulado. La microplaca se centrifugó a 300 × g durante cinco minutos en una centrífuga de cubeta oscilante que se ajustó previamente a 4 °C para el posicionamiento tumoroide, luego se transfirió a una incubadora a 37 °C/CO2 al 5 % durante una hora para iniciar la formación de gel.
Rendimiento del ensayo de invasión tumoral
Una vez que se completó la formación del gel, la microplaca se transfirió a BioSpa 8, donde el software se programó de tal manera que la microplaca se transfirió automáticamente a Cytation 5 para obtener imágenes fluorescentes y de campo claro de los pocillos cada doce horas durante el período de incubación de siete días. La Tabla 1 enumera las configuraciones utilizadas para realizar la captura de imágenes automatizada de cada pocillo de muestra.
Tabla 1. Parámetros de imágenes de invasión tumoroide en 3D automatizados.
| Parámetros de imagen | |
| Canal de imágenes de campo claro | Estructura invasora 3D completa |
| Canal de imágenes GFP | Células U-87 que expresan GFP |
| Canal de imágenes RFP | Fibroblastos que expresan RFP |
| Canal de imágenes CY5 | Células LN-229 teñidas con colorante CellTracker Deep Red |
| Objetivo | 4x |
| Montaje | 3 filas por 2 columnas |
| Superposición de montaje | Automático para puntadas |
| Z-Stack | 20 Slices |
| Z-Stack Step Size | 53.8 μm (Predeterminado para objetivo 4x) |
Procesamiento de imágenes
Luego se unieron mosaicos de imágenes individuales de cada plano z capturado usando el montaje de tres por dos (Tabla 2).
Tabla 2. Parámetros de unión de imágenes.
| Parámetros de unión de imágenes | |
| Canal de registro | Campo Claro |
| Método de fusión | Mezcla lineal |
| Recortar imagen unida para eliminar rectángulos negros en los bordes | Comprobado |
| Reducir imagen final | Comprobado (52.62%) |
A continuación, se creó una proyección de imagen única a partir de la z-stack unida de 20 cortes (Tabla 3). Se eligió el método de apilamiento de enfoque, que selecciona automáticamente el píxel más enfocado de cada imagen en la pila para incluirlo en la proyección final. Esto permite realizar el análisis más preciso en cada tumoroide invasor en cada punto de tiempo.
Tabla 3. Parámetros de proyección z de la imagen.
| Parámetros de proyección z de la imagen | |
| Canal 1 | Unido[Campo Claro] |
| Método | Superposición de enfoque |
| Tamaño del filtro máximo | 11 px |
| Cortes en Z incluidos | 1 a 20 |
| Canales 2 a 4 | Unido[GFP], Unido[RFP], Unido[CY5] |
| Método (Canales 2 a 4) | Usar la configuración del canal 1 |
Análisis de invasión tumoral
Los criterios de análisis celular de máscara primaria (Tabla 4) se aplicaron utilizando el canal de campo claro para colocar automáticamente máscaras de objetos alrededor de toda la estructura invasora en cada imagen final. También se aplicaron criterios de análisis celular de máscara secundaria (Tabla 5) para colocar una máscara adicional alrededor de las células no invasivas que permanecían dentro de la estructura tumoroide esférica original.
Tabla 4. Parámetros de análisis de máscara primaria.
| Parámetros de análisis de máscara primaria | |
| Canal | ZProj[Unido[Campo Claro]] |
| Límite | 25.000 |
| Background | Claro |
| Dividir objetos tocados | No controlado |
| Rellenar agujeros en máscaras | Controlado |
| Tamaño mínimo de objeto | 300 μm |
| Tamaño máximo de objeto | 2.000 μm |
| Incluir objetos de borde principal | Controlado |
| Analizar toda la imagen | Controlado |
| Opciones de detección avanzadas | |
| Diámetro de la bola rodante | 2,000 |
| Fuerza de suavizado de imagen | 0 |
| Evaluar fondo en | 5% de los píxeles más bajos |
| Métrica de análisis | |
| Métrica de interés | Área de objetos |
Tabla 5. Parámetros de análisis de máscara secundaria.
| Parámetros de análisis de máscara secundaria | |
| Canal | ZProj[Unido[Campo Claro]] |
| Medida dentro de una máscara secundaria | Incluir máscara principal |
| Reducir máscara primaria | 1 μm |
| Límite | 12.000 |
| Liso | 7 |
| Rellenar agujeros en la máscara | No contolado |
| Métricas de análisis | |
| Métrica de interés | Objeto Aera_2[ZProj[Unido[Campo Claro]]] |
Resultados y discusión
Procesamiento de imágenes sin procesar antes del análisis celular
Durante el período de incubación de siete días, los tumoroides U-87/fibroblastos desinhibidos continuaron aumentando de tamaño, así como invadiendo la matriz proteica. Para garantizar que toda la estructura invasora, incluidos los invadopodios, se capturara en los ejes X e Y, independientemente del tamaño, se tomaron múltiples imágenes en un solo plano z en un formato de montaje (Tabla 1). La Figura 1 ilustra cuatro de las seis imágenes que fueron capturadas que contienen porciones de la estructura invasora.
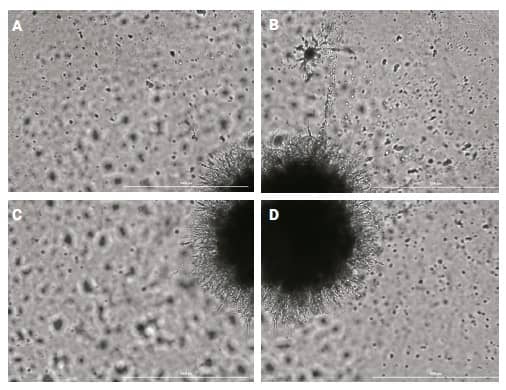
Figura 1. Imágenes de campo claro de U-87/fibroblastos 4x que muestran (A a D) cuatro mosaicos de imágenes que contienen el tumoroide invasor de interés.
A medida que avanza la invasión, las proteínas y las células invaden múltiples planos dentro del eje Z. Por lo tanto, las imágenes se tomaron automáticamente en un rango de alturas z para que todas las partes de la estructura general estuvieran enfocadas en las imágenes finales proyectadas en z (Figuras 2A a 2D).
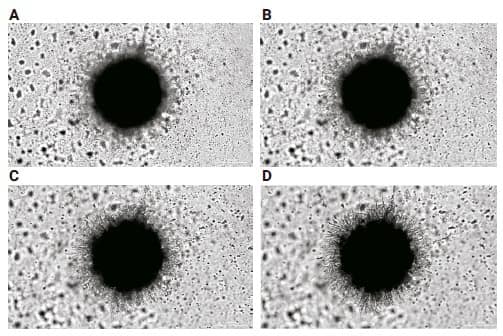
Figura 2. Imágenes de campo claro de U-87/fibroblastos 4x que muestran (A a D) cuatro de las veinte imágenes capturadas en múltiples planos z como parte de la z-stack y combinadas en la proyección z final.
Obtención de imágenes de tipos de células cocultivadas individuales U-87/fibroblastos
Además de las imágenes de campo claro, los mosaicos de montaje y las capas apiladas en z también se capturaron utilizando los canales GFP y RFP. Las imágenes superpuestas rastrean la invasión de toda la estructura (Figura 3).
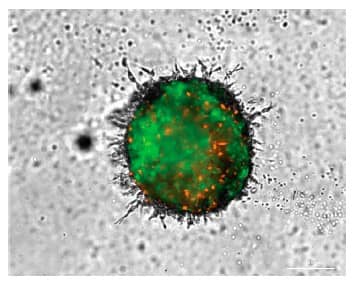
Figura 3. Imagen proyectada en z superpuesta de campo claro y fluorescente del modelo de células cultivadas conjuntamente con U-87/fibroblastos.
Al ver la señal fluorescente emitida por cada tipo de célula cocultivada, en comparación con la señal de campo claro de toda la estructura, se puede observar la migración de células individuales. En el caso del modelo de fibroblastos/U-87, la imagen de campo claro muestra extensos invadopodios que se extienden desde el tumoroide de propagación original (Figura 4A). A partir de la imagen de GFP en la Figura 4B, es evidente que las células U-87 que expresan GFP siguen la invasión de invadopodios en la matriz. Esto contrasta con la imagen de RFP en la Figura 4C, lo que confirma que los fibroblastos que expresan RFP exhiben poca o ninguna capacidad migratoria dentro de este modelo experimental.
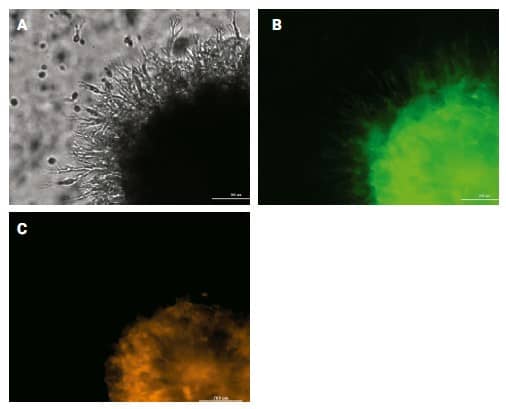
Figura 4. Confirmación de invasión por tipos de células individuales. (A) Invasión tumoroide total a través de canales GFP, RFP y de campo claro superpuestos. Invasión celular individual por (B) células U-87 que expresan GFP, o (C) células de fibroblastos que expresan RFP. Imágenes 4x con zoom digital, montaje 2 × 3, z-stack de 20 planos.
Captura de imagen cinética
Finalmente, las imágenes proyectadas en z se capturan cinéticamente cada 12 horas para observar los cambios fenotípicos en los tumoroides cocultivados con U-87/fibroblastos después del tratamiento con concentraciones variables de 17-AAG. Los tumoroides desinhibidos, como era de esperar, demuestran una naturaleza altamente invasiva4, mostrando un aumento en el tamaño del cuerpo del tumoroide, así como un aumento dramático en los invadopodios (Figura 5).
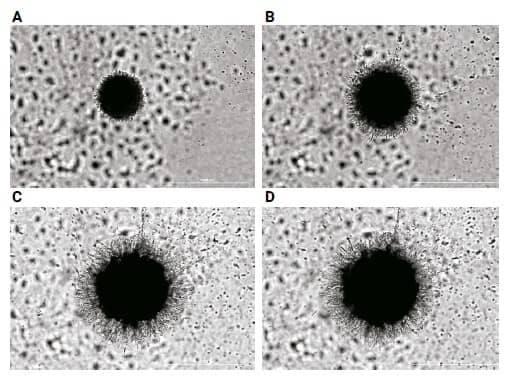
Figura 5. Potencial invasivo tumoroide U-87/fibroblasto a lo largo del tiempo, imágenes de campo claro 4x, montaje 2 × 3, z-stack de 20 planos. (A) 0 horas; (B) 48 horas; (C) 120 horas; (D) 168 horas.
LN-229/Análisis tumoroide de fibroblastos
También se realizaron imágenes de tumoroides LN-229/fibroblastos de la misma manera que para los tumoroides U-87/fibroblastos. Esto se hizo para comparar las diferencias en el crecimiento y la producción de invadopodios entre los tipos de células GBM conocidas por ser altamente invasivas (U-87) y aquellas con capacidad menos invasiva (LN‑229).5 Se observaron cambios en la estructura 3D completa utilizando el campo claro superpuesto. e imágenes fluorescentes (Figura 6A), mientras que la invasión de fibroblastos o LN-229 individual fue monitoreada por la señal capturada con los canales CY5 o RFP individuales, respectivamente (Figuras 6B y 6C).
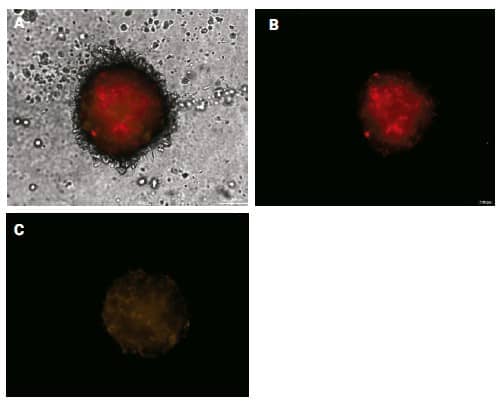
Figura 6. Imágenes del modelo de células cultivadas conjuntamente con LN-229/fibroblastos. (A) Invasión tumoroide total a través de canales CY5, RFP y de campo claro superpuestos. Invasión celular individual por (B) células LN-229 teñidas con CellTracker Deep Red; o (C) células de fibroblastos que expresan RFP. Imágenes 4x con zoom digital, montaje 2 × 3, z-stack de 20 planos.
Al comparar las imágenes cinéticas de campo claro de los dos tipos de células cocultivadas, es evidente que los tumoroides LN-229/fibroblastos se propagan con el tiempo, como se ve por el aumento en el tamaño del esferoide (Figura 7), pero no exhiben las propiedades invasivas claramente demostradas. por tumoroides U-87/fibroblastos durante el mismo período de incubación (Figura 5).
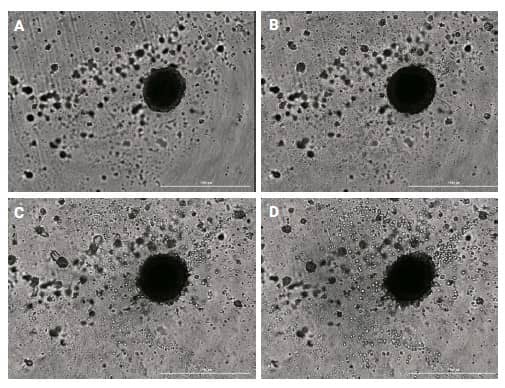
Figura 7. Potencial invasivo tumoroide de LN-229/fibroblastos a lo largo del tiempo, imágenes de campo claro 4x, montaje 2 × 3, z-stack de 20 planos. (A) 0 horas; (B) 48 horas; (C) 120 horas; (D) 168 horas.
Análisis celular basado en Agilent BioTek Gen5 Image+ de invasión tumoroide
Además de las evaluaciones cualitativas de los cambios fenotípicos tumoroides, también se llevó a cabo la cuantificación de la extensión de la invasión utilizando las imágenes de campo claro proyectadas en z cosidas. Se realizaron dos análisis celulares separados después del tratamiento con la titulación de 17-AAG. En el primero, las principales capacidades de análisis celular disponibles en Gen5 Image+ y los criterios enumerados en la Tabla 4 utilizaron cambios en la señal de campo claro dentro de la imagen entre los píxeles que contienen celular y el fondo para colocar máscaras de objetos detalladas alrededor de la estructura 3D invasora completa, a pesar del nivel. de tratamiento compuesto (Figura 8).
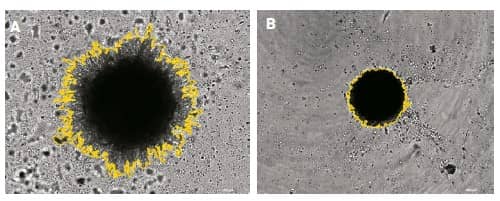
Figura 8. Enmascaramiento de objeto tumoroide invasor. Imágenes ampliadas de campo claro 4x de tumoroides U-87/fibroblastos, montaje 2 × 3, z-stack de 20 planos. (A) tratamiento con 17-AAG 0 μM y (B) tratamiento con 17-AAG 10 μM. Máscaras primarias alrededor de células e invadopodios.
Luego se calculó el área cubierta por todo el tumoroide para cada imagen capturada a lo largo del tiempo. Los valores de los puntos de tiempo posteriores se dividieron luego por el valor del área calculado en el tiempo 0 para el tumoroide específico para normalizar los resultados y tener en cuenta la variabilidad en el tamaño del tumoroide después de la distribución y agregación de células. Luego se trazaron las proporciones de área (eje Y) con respecto al tiempo (eje X) (Figura 9).
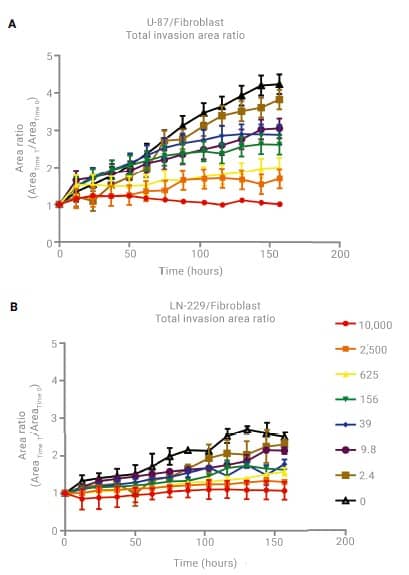
Figura 9. Proporciones de área tumoroide total cinética. Proporciones de área representadas para (A) U-87/tumoroides de fibroblastos, o (B) LN-229/tumores de fibroblastos después de tratamientos de 0 a 7 días con concentraciones de 17-AAG que oscilan entre 10.000 y 0 nM. Relación de área calculada mediante la siguiente fórmula: (Areatime T/Areatime 0).
A partir de los resultados que se observan en la Figura 9, es evidente que tanto los tumoroides U-87/fibroblastos como los LN-229/fibroblastos se propagan dentro de la matriz de Matrigel cuando no se encuentran obstaculizados durante el período de incubación de siete días. También se puede ver que el compuesto 17-AAG es capaz de limitar, o incluso detener por completo, el crecimiento tumoral de una manera dependiente de la dosis, como se esperaba. para la estructura 3D completa exhibida por los tumoroides U-87/fibroblastos en comparación con aquellos en los que las células LN-229 se cocultivan con fibroblastos. Los aumentos en el área cubierta por todo el tumoroide son aproximadamente 2 veces mayores con el tiempo para los tumoroides cultivados con células U-87 (relación: 4,4) en comparación con los cultivados con células LN-229 (relación: 2,4).
Análisis celular basado en Agilent BioTek Gen5 Image Prime de invasión tumoroide
También se realizó un segundo análisis celular utilizando las capacidades de análisis celular primario y secundario disponibles en Gen5 Image Prime y los criterios enumerados en la Tabla 5, para medir el área cubierta únicamente por células no invasoras dentro del tumoroide. Para determinar la capacidad metastásica de los modelos 3D in vitro, tanto sin inhibición como tras el tratamiento, es importante distinguir entre el área cubierta por células dentro del tumoroide original y la cubierta por invadopodios. Como las células de propagación no invasivas más densamente empaquetadas aparecen más oscuras en comparación con los invadopodios en una imagen de campo claro (Figura 10A), este cambio adicional en la señal dentro de la máscara original permite la colocación de una máscara secundaria para excluir las áreas invasoras de cada tumoroide y separar el área. cubierto por las dos porciones de toda la estructura 3D (Figura 10).
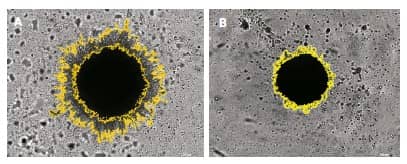
Figura 10. Enmascaramiento de objetos del área tumoroide invasora y principal. Montaje 2 × 3 de campo claro ampliado 4x, imágenes de z-stack de 20 planos de (A) tumoroides U-87/fibroblastos desinhibidos; o (B) tumoroides LN-229/fibroblastos no inhibidos. Máscaras primarias exteriores alrededor de las células y los invadopodios, y máscaras secundarias interiores alrededor de la estructura celular no invasora solamente.
Al aplicar la máscara secundaria alrededor de las porciones no invasoras del tumoroide, las diferencias en las cualidades invasivas de los dos tipos de células GBM se vuelven claras.
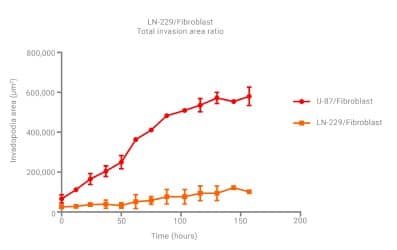
Figura 11. Áreas cinéticas de invadopodios cinéticos tumoroides cocultivados con U-87 y LN-229 sin inhibir cinéticamente. Área de invadopodios calculada a lo largo del tiempo para tumoroides U-87/fibroblastos y LN-229/fibroblastos no tratados. Área de invadopodios calculada mediante la siguiente fórmula: (Total Areatime T – Noninvading Areatime T).
El área cubierta por invadopodios para tumoroides U-87/fibroblastos aumenta dramáticamente durante el período de incubación de siete días. Los tumoroides LN-229/fibroblastos, en comparación, muestran un pequeño aumento en los invadopodios durante el mismo tiempo (Figura 11). Este análisis dual, por lo tanto, tiene el potencial de determinar no solo la rapidez con la que se propagan las células tumoroides, sino también la naturaleza invasiva del modelo celular.
Conclusión
La aplicación de capacidades avanzadas de imágenes celulares, como la toma de múltiples imágenes en un solo plano z, así como en una z-stack, permite capturar enfocadas todas las partes de una estructura tumoroide celular invasora en 3D. Cuando se unen y se proyectan en una imagen final, se puede realizar un análisis preciso. La capacidad de ver tumoroides completos a través de imágenes de campo claro, así como tipos de células individuales a través de imágenes fluorescentes, también permite determinar las propiedades invasivas de cada tipo de célula cocultivada dentro del modelo celular incluido. La inclusión del análisis celular sin etiquetas, realizado en imágenes de campo claro, puede cuantificar no solo las cualidades de propagación, sino también la naturaleza invasiva específica de los modelos celulares 3D in vitro cuando no están inhibidos y en respuesta a las moléculas de prueba.
Referencias
- Furnari, F. B. et al. Growth Suppression of Glioma Cells by PTEN Requires a Functional Phosphatase Catalytic Domain. Proc. Natl. Acad. Sci. USA 1997, 94(23), 12479–84.
- McFall, T. B. Identification of HCMV UL97 in GBM Cell Lines and a Possible Role for Ganciclovir. Master’s Dissertation, Northern Michigan University, Marquette, MI, 2014.
- Beckner, M. E. et al. Identification of ATP Citrate Lyase as a Positive Regulator of Glycolytic Function in Glioblastomas. Int. J. Cancer. 2010, 126(10), 2282–95.
- Formolo, C. A. et al. Secretome Signature of Invasive Glioblastoma Multiforme. J. Proteome Res. 2011, 10(7), 3149–59.
- Mallawaaratchy, D. M. et al. Membrane Proteome Analysis of Glioblastoma Cell Invasion. J. Neuropathol. Exp. Neurol. 2015, 74(5), 425–41.