Un elemento esencial de la biología celular y molecular es la capacidad de cuantificar el ADN en un gran número de muestras con una sensibilidad que permite la determinación de pequeñas cantidades de muestra. Esta nota de aplicación describe un método para cuantificar dsDNA utilizando un lector multimodo Agilent BioTek Synergy Neo2.
Uso de un lector multimodo Agilent BioTek Synergy Neo2 para cuantificar el ADN en solución
Autor: Paul Held, PhD. Agilent Technologies, Inc
Resumen
Un elemento esencial de la biología celular y molecular es la capacidad de cuantificar el ADN en un gran número de muestras con una sensibilidad que permite la determinación de pequeñas cantidades de muestra. Esta nota de aplicación describe un método para cuantificar dsDNA utilizando un lector multimodo Agilent BioTek Synergy Neo2.
Introducción
Muchas técnicas de biología celular y molecular requieren la capacidad de cuantificar dsDNA en un gran número de muestras a sensibilidades que solo requieren una pequeña cantidad de la muestra total. El aislamiento de plásmidos de cultivos bacterianos, el ADN genómico de células de mamíferos, la síntesis de ADNc para la producción de bibliotecas y la cuantificación de productos de PCR con fines de diagnóstico requieren la cuantificación directa de dsDNA. Además, muchos estudios bioquímicos que involucran la cinética de crecimiento de cultivos celulares o estudios del ciclo celular requieren normalización por contenido de ADN.
El método más comúnmente utilizado para la determinación de la concentración de ácido nucleico es la determinación de la absorbancia a 260 nm (A260) según lo descrito por Matiatis. Este método, aunque es bastante adecuado para muchas situaciones, puede verse afectado por la interferencia de la absorbancia de las moléculas contaminantes. Muchos de estos contaminantes, que incluyen nucleótidos, ARN, EDTA y fenol, se encuentran comúnmente en las preparaciones de ácidos nucleicos. Como resultado, se han desarrollado varias técnicas de tinción fluorescente para aliviar muchos de los problemas asociados con la absorbancia a 260 nm. Una de esas tinciones es PicoGreen, que, junto con el lector multimodo Agilent BioTek Synergy Neo2, ofrece una alta especificidad, así como una alta sensibilidad para la cuantificación de dsDNA.
Materiales y métodos
El reactivo Quant-iT PicoGreen (número de pieza P11495), el ADN de esperma de salmón (número de pieza 15632011) y el concentrado de tampón TE (100x) (número de pieza BP-1338-1) se adquirieron de Thermo Fisher Scientific. Las microplacas negras sólidas de 96 pocillos (número de pieza 3915) eran de Corning. El ADN de esperma de arenque se diluyó a 2 µg/ml con TE (Tris 10 mM, EDTA 1 mM, pH 7,5) como diluyente. La concentración final se confirmó utilizando una absorbancia de 260 nm. Se realizó una serie de diluciones que oscilaban entre 0,0 y 2000 ng/ml de esperma de arenque purificado utilizando TE y alícuotas de 100 µl pipeteadas en pocillos de microplaca.
Se mezclaron cantidades iguales (100 µL) de reactivo de cuantificación PicoGreen de trabajo y se incubaron durante 10 minutos a temperatura ambiente, protegidas de la luz. El reactivo de trabajo PicoGreen se preparó diluyendo la solución madre concentrada de DMSO PicoGreen, proporcionada en el kit PicoGreen, 1:200 con TE de acuerdo con las instrucciones del kit.2 Después de la incubación, se determinó la fluorescencia usando un lector multimodo Agilent BioTek Synergy Neo2.
Mediciones basadas en métodos
Con mediciones basadas en filtros, el lector usó un filtro de excitación de 485 nm, 20 nm de ancho de banda y un filtro de emisión de 528 nm, 25 nm de ancho de banda junto con un espejo dicroico de corte de 510 nm. Las determinaciones basadas en monocromadores se realizaron con una longitud de onda de excitación de 485 nm y una longitud de onda de emisión de 528 nm, con el ancho de banda predeterminado del lector. El lector se programó para escalar automáticamente la configuración de sensibilidad a la concentración de ADN más alta. Los datos se recopilaron desde la parte superior con un retraso de 0 milisegundos después del movimiento de la placa, 25 lecturas por pocillo.
Los escaneos espectrales de emisión se realizaron fijando la excitación en 485 nm (ancho de banda de 9 nm) y variando las longitudes de onda de emisión en incrementos de 1 nm. Los escaneos de excitación se produjeron fijando la longitud de onda de emisión en 528 nm y variando las longitudes de onda de excitación en incrementos de 1 nm. Todos los anchos de banda se establecieron en 9 nm y la configuración de ganancia de PMT se estableció en 125 usando una velocidad de lectura normal. Las reacciones se determinaron típicamente en una hora.
Resultados y discusión
Los escaneos espectrales fluorescentes de dsDNA (1 µg/mL) teñidos con el reactivo PicoGreen demuestran picos de excitación y emisión a 485 nm y 528 nm respectivamente después de la sustracción en blanco (Figura 1). Estas longitudes de onda se usaron luego para mediciones posteriores con el fin de maximizar la señal de fluorescencia para los experimentos de titulación de ADN.
Figura 1
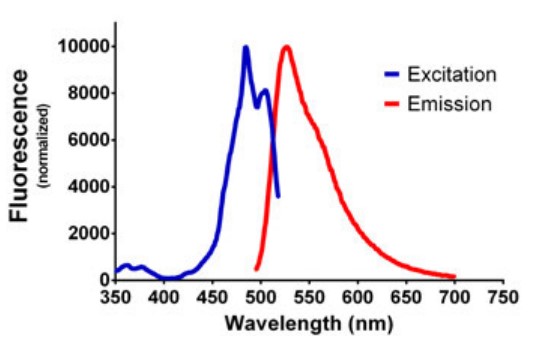
Figura 1. Espectro de emisión y excitación fluorescente de dsDNA (1 µg/mL) teñido con reactivo PicoGreen. Los datos se normalizaron de manera que el valor máximo fue de 10.000 RFU.
La intensidad de la fluorescencia se determinó para concentraciones de ADN que oscilaban entre 0 y 1000 ng/mL. Sobre este rango, la intensidad aumentó de forma lineal. Con el software de reducción de datos Agilent BioTek Gen5, se puede generar un análisis de regresión lineal de mínimos cuadrados con un valor de coeficiente de determinación (R2) de 0,998. El coeficiente de variación promedio (%CV) de los estándares fue inferior al 3%, con la mayor variación en las concentraciones de ADN más bajas analizadas (datos no mostrados). En términos de sensibilidad, se encontró que el ensayo era sensible al nivel de picogramos. Bajo los ajustes de sensibilidad apropiados, se encontró que las concentraciones de ADN tan bajas como 30 pg/mL eran estadísticamente diferentes (P <0.05) del control TE solo, 0 ng/mL.
La cuantificación de dsDNA utilizando las propiedades fluorescentes de PicoGreen, junto con Agilent BioTek Synergy Neo2, permite a los investigadores cuantificar tan solo 6 pg/pocillo (30 pg/ml en un volumen total de 0,2 ml), lo que proporciona una cuantificación fiable de las concentraciones de dsDNA oscilando en cuatro órdenes de magnitud.
Figura 2
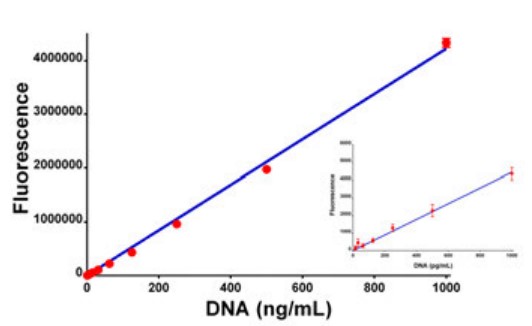
Figura 2. Linealidad de la detección basada en filtros espectrales. La fluorescencia de la titulación de dsDNA incubada con reactivo PicoGreen se realizó utilizando un cubo de filtro 485/528 Agilent BioTek Synergy Neo. La concentración varió de 0,0 a 1 µg/mL de dsDNA con análisis de regresión lineal de mínimos cuadrados. La figura insertada muestra los puntos de datos para las concentraciones más bajas (0 a 1000 pg/mL). Los puntos de datos representan los valores medios de ocho determinaciones en cada concentración.
Además de los cubos de filtro para la detección de fluorescencia, Synergy Neo2 se puede configurar con monocromadores duales para excitación y emisión. Se puede observar una linealidad similar usando los monocromadores para la selección de longitud de onda en comparación con la detección basada en filtros (Figura 3). Se observaron límites de detección similares (30 pg/ml) con los monocromadores y con el sistema óptico basado en filtros.
Figura 3
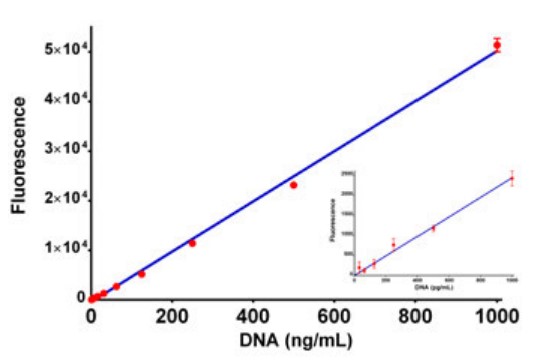
Figura 3. Linealidad de la detección basada en monocromador. La fluorescencia de la titulación de dsDNA incubada con reactivo PicoGreen se realizó utilizando un monocromador Agilent BioTek Synergy Neo 2. La excitación se fijó en 485 nm y la emisión en 528 nm, con un ancho de banda de 20 nm tanto para la excitación como para la emisión. La concentración varió de 0,0 a 1 µg/mL de dsDNA con análisis de regresión lineal de mínimos cuadrados. La figura insertada muestra los puntos de datos para las concentraciones más bajas (0 a 1000 pg/mL). Los puntos de datos representan los valores medios de ocho determinaciones en cada concentración.
A pesar de la menor magnitud en la escala bruta, los monocromadores proporcionan una mejor relación señal/fondo en comparación con los filtros, como se demuestra en la Figura 4. Esto es principalmente el resultado de las mediciones de fondo más bajas producidas por los monocromadores duales.
Figura 4
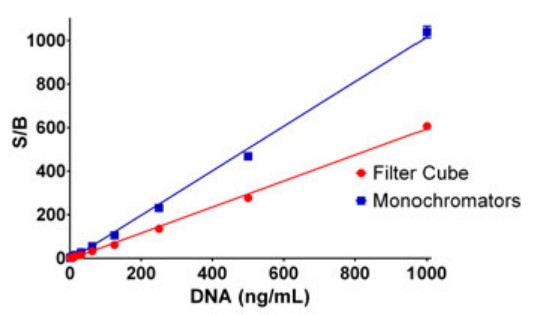
Figura 4. Comparación de las relaciones de señal a fondo de las rutas ópticas basadas en filtros y monocromadores. Se representó gráficamente la relación de cada muestra con la media del blanco (0 µg/mL dsDNA). Los datos representan la media y la desviación estándar de ocho determinaciones
Conclusión
El lector multimodo Agilent BioTek Synergy Neo2 es un lector de microplacas multimodo altamente configurable. Este estudio examinó los sistemas ópticos de detección de fluorescencia basados en filtros y basados en monocromadores. La detección basada en filtros se proporciona con una matriz de cubos de filtro predefinidos que tienen identificación de código de barras. Esto permite al lector asegurarse de que se utiliza la combinación de filtros correcta para el resto fluorescente previsto. Las características espectrales de los filtros y espejos dicroicos empleados maximizan la luz excitatoria sobre el blanco, así como la recuperación de la señal emitida, maximizando la sensibilidad.
El sistema de monocromador dual, aunque da como resultado una señal sin procesar más baja que el sistema de filtro, ofrece una flexibilidad completa en lo que respecta a la selección de longitud de onda, así como al ancho de banda. Además, el diseño de alta resolución de los monocromadores da como resultado una fluorescencia de fondo muy baja, lo que se traduce en una mejor relación señal-blanco (S/B) que la óptica basada en filtros.
Para lograr una sensibilidad óptima, se pueden emprender varias medidas. La eliminación de ciertos contaminantes comunes, como proteínas u orgánicos (por ejemplo, fenol y butanol), dará como resultado CV más ajustados y una señal de fondo más baja, lo que dará como resultado un aumento en la sensibilidad. Se sabe que varios compuestos reducen la señal fluorescente cuando están presentes en un ensayo de ADN PicoGreen. En particular, la presencia de cloruro de sodio 200 nM o cloruro de magnesio 50 mM puede disminuir la señal en aproximadamente 1/3. En los casos en los que solo se deban determinar concentraciones bajas de ADN, se puede utilizar una curva de calibración que utilice concentraciones más bajas. Además, se esperaría que el uso de microplacas que usan plásticos fluorescentes más bajos reduzca el fondo y, por lo tanto, aumente la sensibilidad.
Referencias
- Maniatis T.; Fritsch, E. F.; Sambrook, J. Molecular Cloning A Laboratory Manual, Cold Spring Harbor Laboratory, Cold Springs Harbor, NY; 1982.
- PicoGreen dsDNA Quantitation Reagent and Kit Instruction, Molecular Probes, Eugene, OR; 1996.
- Labarca, C.; Paigen, K. A Simple Rapid, and Sensitive DNA Assay Procedure. Analytical Biochemistry 1980, 102, 344–352.
- Ogura, M. et al. Use of the Fluorescent Dye YOYO-1 to Quantify Oligonucleotides Immobilized on Plastic Plates. BioTechniques 1994, 16, 1032–1033

