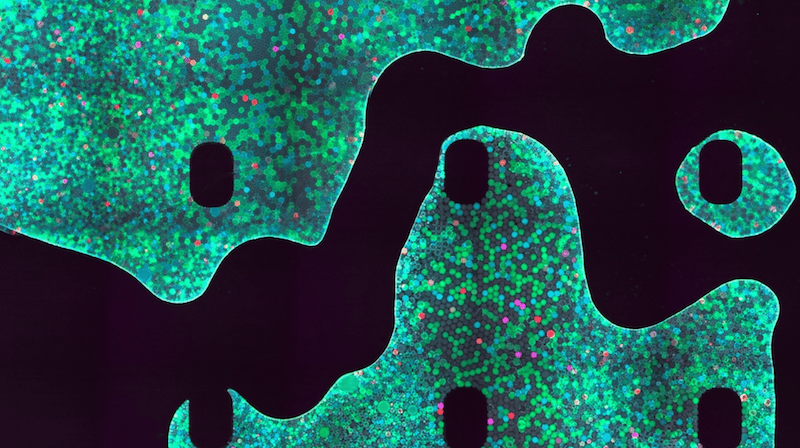Monitoreo de la infección viral de células. La cuantificación de virus implica la determinación del número de virus en un volumen específico. Un método para determinar un título viral es el ensayo de dilución de punto final, que determina una dosis infectiva de cultivo de tejido del 50% (TCID50).
Síntesis
Monitoreo de la infección viral de células. La cuantificación de virus implica la determinación del número de virus en un volumen específico. Un método para determinar un título viral es el ensayo de dilución de punto final, que determina una dosis infectiva de cultivo de tejido del 50% (TCID50). Este método consiste en infectar células de cultivo de tejidos y es útil para virus que no forman placas. Esta nota de aplicación demuestra la capacidad del lector multimodo de obtención de imágenes de células Agilent BioTek Cytation 5 y el software de lectura de microplacas y generación de imágenes Agilent BioTek Gen5 para determinar los niveles de infección del virus de la inmunodeficiencia humana (VIH) con células HeLa en cultivo.
Introducción
La cuantificación viral determina el número de virus en un volumen específico de líquido. Su utilidad es más evidente en la producción de vacunas virales o proteínas recombinantes que utilizan vectores virales como medio para la entrada o propagación celular. Hay varios métodos diferentes de cuantificación de virus; el más notable es el ensayo de placas, donde la infección viral provoca la lisis celular. En este ensayo, una monocapa confluente de células se infecta con virus, lo que provoca la lisis de las células infectadas. Las células circundantes se infectarán y eventualmente se habrá denudado un área más grande de la monocapa celular.
Estas áreas, denominadas placas, se pueden contar para calcular una unidad formadora de placas, que es una medida de la cantidad de virus. El ensayo de formación de focos (FAA por sus siglas en inglés) está relacionado con el ensayo de placas, excepto que utiliza inmunotinción fluorescente con anticuerpos marcados para determinar la infección viral. Este método es útil para los virus que no lisan las membranas celulares como parte de su ciclo de vida. Una modificación de los dos métodos descritos anteriormente es el ensayo de dilución de punto final, que calcula una dosis infectiva de cultivo de tejidos del 50 % (TCID50).
Esto cuantifica la cantidad de virus requerida para matar o infectar el 50% de las células huésped inoculadas, y es similar a una LD50 o IC50 comúnmente determinada durante el desarrollo de fármacos. El VIH es un retrovirus que causa la infección por VIH y el síndrome de inmunodeficiencia adquirida (SIDA).1 El VIH es un lentivirus que infecta a varios tipos de células diferentes y se integra en el genoma celular como parte de su ciclo de crecimiento (Figura 1).2
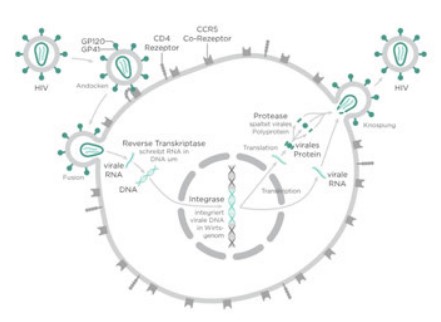 Figura 1. Diagrama esquemático de la infección por lentivirus del VIH en células de mamífero. Imagen proporcionada por cortesía de Thomas Splettstoesser.
Figura 1. Diagrama esquemático de la infección por lentivirus del VIH en células de mamífero. Imagen proporcionada por cortesía de Thomas Splettstoesser.
Materiales y métodos utilizados
Cultivo de células
Las células HeLa se cultivaron en DMEM complementado con suero bovino fetal al 10 % y penicilina-estreptomicina a 37 °C en CO2 al 5 %. Los cultivos se tripsinizaron rutinariamente (0,05 % de tripsina EDTA) al 80 % de confluencia.
Infección vírica
Para los experimentos, las células se sembraron en microplacas de 96 pocillos Corning 3904 de fondo transparente con lados negros a 5.000 células por pocillo. Se permitió que las células crecieran durante la noche, con infección por VIH al día siguiente. Se infectaron diferentes pocillos con titulaciones 1:2 de cepas de virus que expresan HIV-GFP o HIV-mCherry. Después de 48 horas, se aspiró el medio y se fijaron las células con paraformaldehído al 2 % (100 µl) durante la noche.
Tinción
Después de la fijación, la solución de paraformaldehído se eliminó por aspiración y los pocillos se lavaron una vez con PBS. Se añadió solución de DAPI (0,25 µg/mL) y las células se tiñeron durante 15 minutos a temperatura ambiente. A continuación, la solución de DAPI se eliminó por aspiración y las células se lavaron una vez con PBS. A continuación, se añadió PBS (100 µl) a los pocillos para mantener la hidratación durante la obtención de imágenes.
Imágenes
Se tomaron imágenes de los experimentos usando un lector multimodo de imágenes de células Cytation 5 configurado con cubos de luz DAPI, GFP y Texas Red. La cámara utiliza fuentes de luz LED junto con filtros de paso de banda y espejos dicroicos para proporcionar luz de longitud de onda adecuada. El cubo de luz DAPI usa un filtro de excitación 337/50 y un filtro de emisión 447/60, el cubo de luz GFP usa un filtro de excitación 469/35 y un filtro de emisión 525/39, mientras que el cubo de luz Texas Red usa una excitación 585/29 filtro y un filtro de emisión 624/40.
Análisis de imagen
Por lo general, las imágenes de las muestras se tomaron capturando un montaje de imágenes y creando una imagen compuesta unida de un campo de visión más amplio. Los mosaicos de montaje (5 × 4) de imágenes objetivas 4x superpuestas en tres colores se unieron digitalmente utilizando el software Gen5. Se usó el conteo de máscaras de objetos del canal DAPI para identificar núcleos celulares. El análisis de subpoblaciones se utilizó para determinar la intensidad de fluorescencia media de los canales Texas Red y GFP como un medio para evaluar las células positivas para mCherry y GFP, respectivamente.
Las máscaras de objetos de núcleos celulares se delinearon utilizando un umbral de 12.000 con una exclusión de tamaño mínimo de 5 µm y máximo de 50 µm. El análisis de subpoblaciones definió pozos positivos para GFP y mCherry con umbrales de fluorescencia media de GFP y TR de 11 000 y 7 000, respectivamente.
Citometría de flujo
Las células HeLa infectadas con las mismas titulaciones de VIH marcado con GFP que las utilizadas para la formación de imágenes se fijaron y tiñeron y luego se recolectaron para citometría de flujo. El control negativo se usó para establecer la activación positiva de GFP. A continuación, se utilizó la misma puerta para todos los análisis de citometría de flujo posteriores.
Resultados y discusión
La infección viral se puede demostrar con imágenes de fluorescencia. Como se muestra en la Figura 2, la infección de células HeLa con virus etiquetados con GFP produce una fluorescencia verde significativa. Del mismo modo, la infección de células con virus etiquetados con mCherry también se puede detectar en relación con el control negativo no infectado (Figura 3).
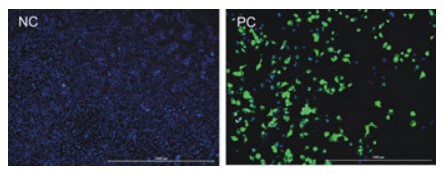 Figura 2. Comparación de células HeLa infectadas y no infectadas con VIH-GFP. Se capturaron 4x digitales en los canales DAPI, Texas Red y GFP y se superpusieron las imágenes.
Figura 2. Comparación de células HeLa infectadas y no infectadas con VIH-GFP. Se capturaron 4x digitales en los canales DAPI, Texas Red y GFP y se superpusieron las imágenes.
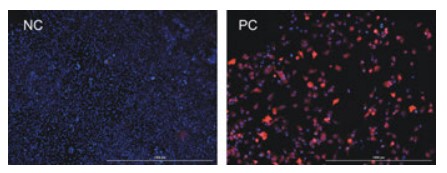
Figura 3. Comparación de células HeLa infectadas y no infectadas con VIH-mCherry. Se capturaron 4x digitales en los canales DAPI, Texas Red y GFP y se superpusieron las imágenes.
Estos datos también demuestran la fidelidad de la señal ya que ambos experimentos involucraron la imaginación de tres colores. Solo las células infectadas con el virus etiquetado con GFP exhibieron fluorescencia verde (Figura 2), y solo aquellas células infectadas con el virus etiquetado con mCherry tenían señal en el canal Texas Red (Figura 3). Los controles negativos no infectados para cualquiera de los experimentos solo tenían tinción DAPI.
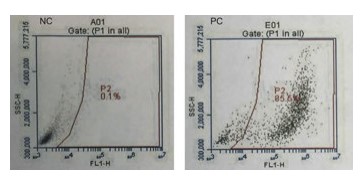
Figura 4. Comparación del análisis de diagrama de dispersión del citómetro de flujo de células HeLa infectadas y no infectadas con GFP de VIH. El resultado de la citometría de flujo de las células infectadas teñidas con DAPI se representó gráficamente de tal manera que la intensidad de la fluorescencia verde se representó en el eje, mientras que la fluorescencia azul se representó en la abscisa.
La presencia del virus del VIH tiene un efecto significativo en el número total de células HeLa presentes en el momento de la fijación (Figura 5). Los recuentos de células de núcleos teñidos realizados con diferentes títulos de virus muestran una disminución en el número de núcleos de células HeLa teñidas con el aumento de la carga viral. El análisis basado en imágenes de células infectadas con HIV-GFP o HIV-mCherry muestra una disminución de 41 000 células en el control no infectado a 6000 células en los pocillos con la mayor concentración de virus.
Asimismo, los recuentos de células de citometría de flujo muestran un patrón similar con la excepción del control negativo, que informa un recuento de células un 50% superior. Lo más probable es que la diferencia entre los dos sistemas se atribuya al empaquetamiento compacto de células confluentes, lo que dificulta que el análisis de imágenes distinga núcleos separados a densidades muy altas.
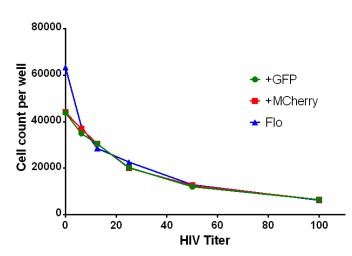
Figura 5. Recuentos de células de pozo completo de células HeLa infectadas por VIH. Las máscaras de objetos de núcleos teñidos con DAPI se contaron a partir de células HeLa infectadas con VIH GFP o virus VIH-mCherry. En cada caso, se realizó un análisis de pocillo completo en imágenes de montaje que posteriormente se unieron con el software lector de microplacas y generador de imágenes Agilent BioTek Gen5. También se realizó una comparación usando recuentos de células de un citómetro de flujo de células infectadas con HIV GFP tripsinizadas realizadas en paralelo.
Usando la tinción DAPI para identificar una máscara nuclear para el análisis de imágenes, se puede rastrear el alcance de la infección mediante el análisis de subpoblaciones. Los objetos definidos por la máscara de umbral principal como núcleos pueden luego someterse a un análisis de subpoblación basado en otros colores fluorescentes. Cuando se trazan los porcentajes de células positivas para GFP, se observó un aumento en el recuento de células positivas que se corresponde con la carga viral de HIV-GFP (Figura 6). Las células infectadas con mCherry-tagged HIV no muestran ningún aumento cuando se seleccionan para GFP.
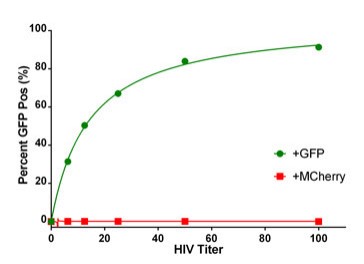
Figura 6. Porcentaje de células HeLa infectadas con VIH positivas para GFP. Las células HeLa se infectaron con una variedad de títulos virales con el virus HIV-GFP y HIV-mCherry. El número total de células se determinó usando una máscara nuclear de núcleos teñidos con DAPI y análisis de subpoblaciones de células que expresan GFP. Los datos se informaron como un porcentaje de células positivas para GFP.
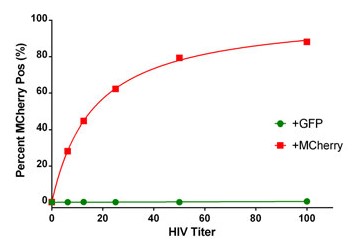
Figura 7. Porcentaje de células HeLa infectadas con VIH positivas para mCherry. Las células HeLa se infectaron con una variedad de títulos virales con el virus HIV-GFP y HIV mCherry. El número total de células se determinó utilizando una máscara nuclear de núcleos teñidos con DAPI y análisis de subpoblaciones de células que expresan mCherry. Los datos se informaron como un porcentaje de células positivas para mCherry.
Una comparación entre el porcentaje calculado de células positivas para GFP de VIH mediante análisis de imágenes o citometría de flujo muestra una muy buena concordancia entre los dos métodos (Figura 8). El TCID50 para la titulación del virus mediante citometría de flujo fue de 11,95, mientras que los datos de análisis de imágenes tienen un TCID50 de 12,68. Se esperarían resultados similares con mCherry (datos no mostrados).
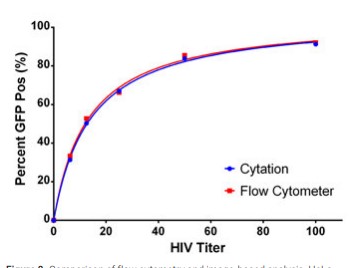
Figura 8. Comparación de citometría de flujo y análisis basado en imágenes. Las células HeLa infectadas con varios títulos de HIV-GFP se analizaron para la expresión de GFP usando citometría de flujo o análisis basado en imágenes de microscopía digital. Los datos se expresaron como el porcentaje positivo en cada título viral.
Conclusión sobre el monitoreo de la infección viral de células
Estos datos demuestran la capacidad del lector multimodo de imágenes celulares Agilent BioTek Cytation 5 y el software lector de microplacas y generador de imágenes Agilent BioTek Gen5 para rastrear los aumentos en la infección por virus cuando se utilizan partículas virales marcadas con fluorescencia. Usando análisis de tres colores, se demostró que el aumento de la señal como resultado de la infección por el virus es específico de la etiqueta adherida al virus.
En estos experimentos, se obtuvieron imágenes de montaje de todo el pozo y los mosaicos separados se unieron en un solo archivo de datos. Esta práctica elimina el conteo duplicado de la misma celda en imágenes adyacentes. Además, el uso de imágenes de montaje unidas de todo el pozo para el análisis proporciona mediciones confiables de todo el pozo sin la necesidad de extrapolar en función del área de superficie del pozo examinada con una sola imagen.
El recuento total de células y el porcentaje de células VIH positivas se utilizaron como medio para comparar el análisis basado en imágenes con la citometría de flujo. Con densidades de células muy altas, la proximidad de los núcleos de las células dificulta los recuentos de células precisos, lo que da como resultado una subestimación del número real. Sin embargo, cuando la densidad celular es tal que las células son discretas, el análisis de imágenes arroja valores similares a los observados con la citometría de flujo. Mediante el análisis de subpoblaciones, se encontró que el porcentaje de células VIH positivas era idéntico al de la citometría de flujo o al del análisis de imágenes.
Fuente: Agilent Biotek – Application Note – Microbiology – Monitoring Viral Infection of Mammalian Cells Using Digital Fluorescence Microscopy
References
- Weiss, R. A. How does HIV cause AIDS?. Science 1993, 260(5112), 1273–9. Bibcode:1993Sci…260.1273W. doi:10.1126/science.8493571.PMID 8493571
- Smith, J. A.; Daniel, R. Following the path of the virus: the exploitation of host DNA repair mechanisms by retroviruses. ACS Chem. Biol. 2006, 1(4), 217–26 doi:10.1021/cb600131q. PMID 17163676.